高歌研究组2022年发表于《自然·生物技术》(Nature Biotechnology)杂志的研究成果《基于图耦联的单细胞多组学数据整合和调控推断》(Multi-omics single-cell data integration and regulatory inference with graph-linked embedding),近日经《基因组蛋白质组与生物信息学报》(Genomics, Proteomics and Bioinformatics,简称GPB)评选,入选2022年度“中国生物信息学十大进展”。
单细胞组学技术近年来蓬勃发展,已成为研究细胞功能及其背后的基因调控机制的重要手段,目前许多不同组学已能在单细胞精度下进行检测,每种组学反应的是细胞状态的一个侧面,为了更准确地整合不同组学数据,全面刻画细胞内的基因调控状态和调控机制,高歌团队开发了基于图耦联的计算模型GLUE,该模型采用变分自编码器(Variational AutoEncoder, VAE)学习细胞的降维表示,由于特征空间的差异,不同组学的细胞需要使用不同的自编码器,为了在不同组学的自编码器之间建立关联,GLUE提出了图耦联(graph-linking)策略,将组学特征间的先验调控知识组织成一个引导图(guidance graph),有效将不同组学的自编码器关联起来,保证降维空间具有“语义一致性”(图 1)。GLUE实现了很高的整合精度、鲁棒性和可扩展性,可以自然地支持整合任意数量、调控方向的组学数据,并同时进行调控推断。
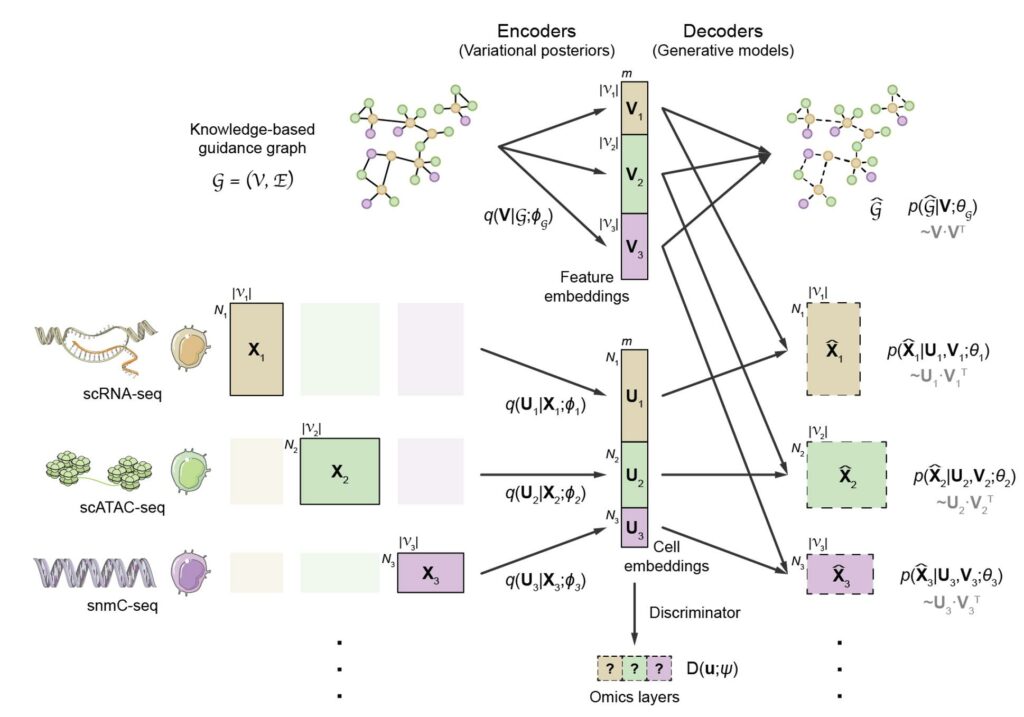
图1:GLUE模型的结构示意图
中国生物信息学“十大”系列是由《基因组蛋白质组与生物信息学报》于2018年发起组织评选,旨在推动我国生物信息学的学科发展和创新研究,充分展示和宣传我国生物信息学领域的重大研究成果,高歌研究组人类lncRNA图谱和单细胞转录组细胞检索方法Cell BLAST分别曾入选2019年度“中国生物信息学十大数据库”和2020年度“中国生物信息学十大进展”。
工具链接:
https://github.com/gao-lab/GLUE
原文信息:
Cao, Z.-J. & Gao, G. Multi-omics single-cell data integration and regulatory inference with graph-linked embedding. Nat. Biotechnol. 2022;40:1458-66. PMID: 35501393