近年来单细胞转录组测序的迅猛发展,为细胞功能和基因调控网络等重要生物学问题的研究提供了强大的技术支持。在单细胞转录组数据的相关研究中,研究者通常会先对细胞进行注释,如鉴定细胞类型、细胞分化阶段等,然而,常用的注释手段较为繁琐,且无法保证不同数据集间的可比性。随着单细胞转录组数据逐渐积累,用现有数据集作为参考(reference),来注释新测序的细胞成为一种潜在的解决方案。
利用现有数据意味着需要在不同单细胞转录组数据之间进行比较,会遇到一个广义上称为批次效应(batch effect)的问题,导致批次效应的具体原因有非常多,包括采用不同的单细胞转录组建库技术带来的转录本捕捉效率和序列偏好差异、不同实验批次的操作差异、不同测序批次的测序深度差异、不同物种表达调控的差异,甚至是不同生物信息学分析的流程差异等等。若无法将数据集之间的批次效应消除,会导致跨数据集比较的准确性大打折扣,影响对现有数据的利用。
对抗生成网络(GAN)是近年来深度学习领域的一个重要发展,GAN通过让“生成器”和“判别器”相互对抗,促使“生成器”生成和目标分布无法区分的样本,在图像、文本生成等领域取得了许多重大突破。得益于对抗学习方式强大的分布拟合能力,在生成学习以外也被成功应用于领域适应(domain adaptation)1等问题,具有解决单细胞领域批次效应的潜力。
因此,我们开发了一个基于深度对抗学习模型的单细胞转录组数据检索和注释的新方法Cell BLAST,以及具备高质量注释的单细胞转录组参考数据库ACA,为有效利用现有数据进行细胞注释和跨数据集研究提供新的工具和资源。
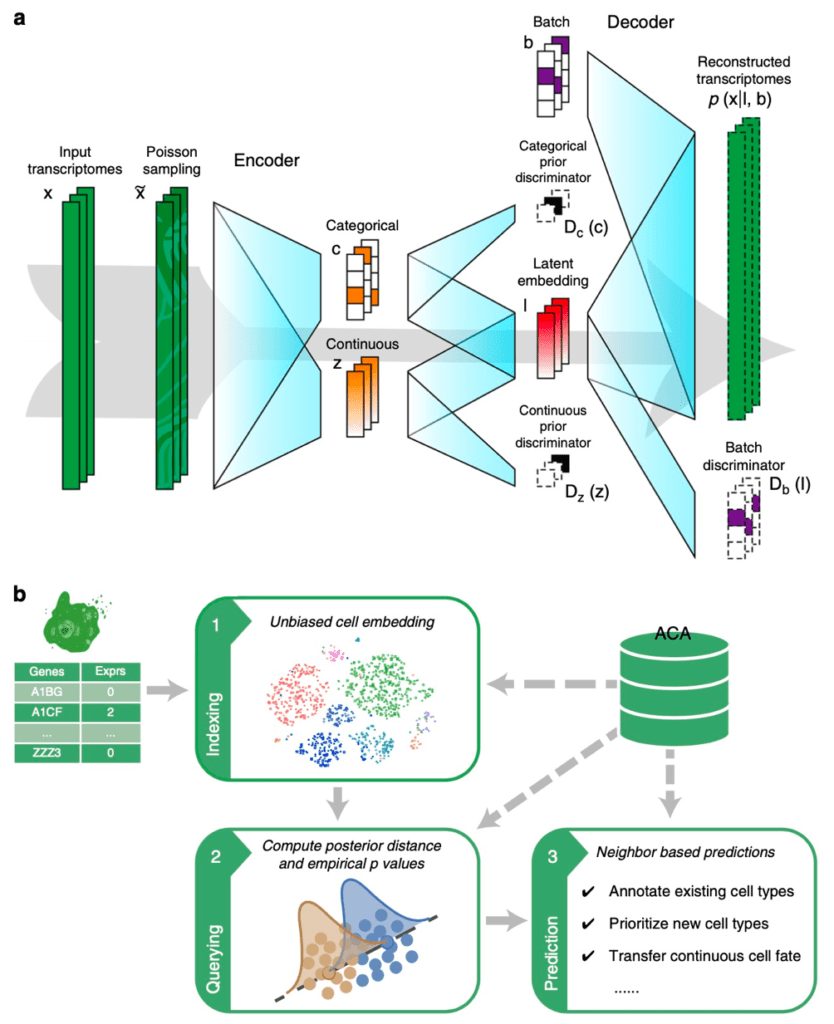
图1:Cell BLAST使用的模型结构和检索流程。(a) Cell BLAST使用的模型结构;(b) Cell BLAST的检索流程。
类比于生物序列研究中的BLAST算法,Cell BLAST可以在若干reference数据集中检索与用户提供的query细胞最相似的细胞,并借助这些相似细胞在数据库中的注释信息,对query细胞的注释信息进行推断。然而,在细胞检索过程中,数据集之间的批次效应会显著影响结果的可靠性,为解决这一问题,Cell BLAST使用对抗自编码器(Adversarial Autoencoder)进行转录组数据降维,结合领域对抗学习的策略来消除数据集间的批次效应(图1a),取得了优于当前其他批次效应校正工具的效果(图2a)。此外,我们提出了一个基于模型后验分布的、更为准确的细胞相似性度量NPD用于细胞检索,在设计上考虑了单细胞转录组观测本身所具有的不确定性。Cell BLAST还会根据NPD的经验分布给出检索结果的P-value。评测实验显示,Cell BLAST在细胞类型鉴定方面,相比其他细胞检索工具拥有更高的准确性(图2b, c)。通过梯度回传的方式,我们进一步验证了Cell BLAST模型正确捕捉到了细胞类型的marker基因,具有一定的可解释性。
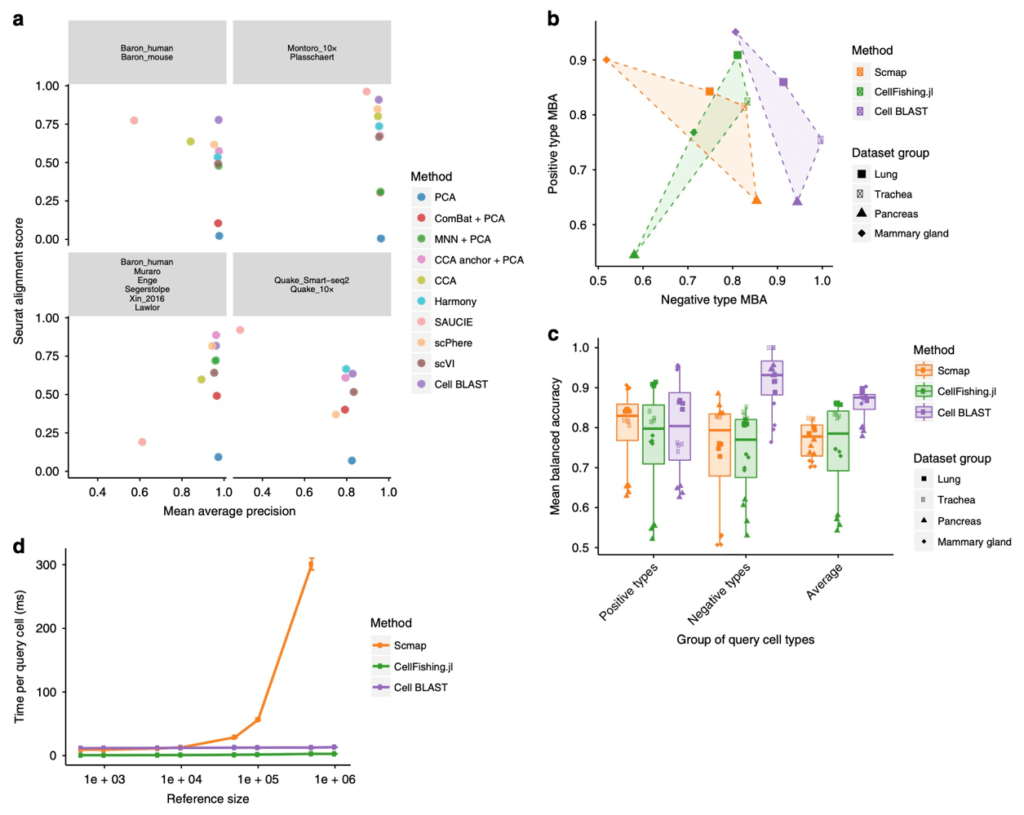
图2:Cell BLAST性能测试。(a) 批次效应消除效果比较;(b) 基于检索的细胞类型预测在阳性query(reference中存在的细胞类型)和阴性query(reference中不存在的细胞类型)上的准确度;(c) 基于检索的细胞类型预测总体性能比较;(d) 检索速度比较。
除细胞类型鉴定外,Cell BLAST能灵敏地发现参考数据集中不存在的细胞类型。2018年Nature发表的两篇背靠背单细胞转录组研究同时鉴定出呼吸道稀有细胞类型ionocyte2, 3。我们以这两篇研究的数据集为例,将其中一个数据集中的ionocyte去除后作为reference,用来注释另一个数据集,Cell BLAST灵敏地发现了这类细胞的特殊性,并将其reject,而没有错误地预测为其他已知的细胞类型(图3a-c);除此以外,我们利用人类和小鼠造血干细胞分化的数据集4, 5验证了Cell BLAST还能用于跨物种注释连续细胞状态这一更具挑战性的任务(图3d-f),相比其他现有工具,Cell BLAST跨物种预测的细胞分化命运,与已知的命运决定基因的表达水平具有更高的相关性。
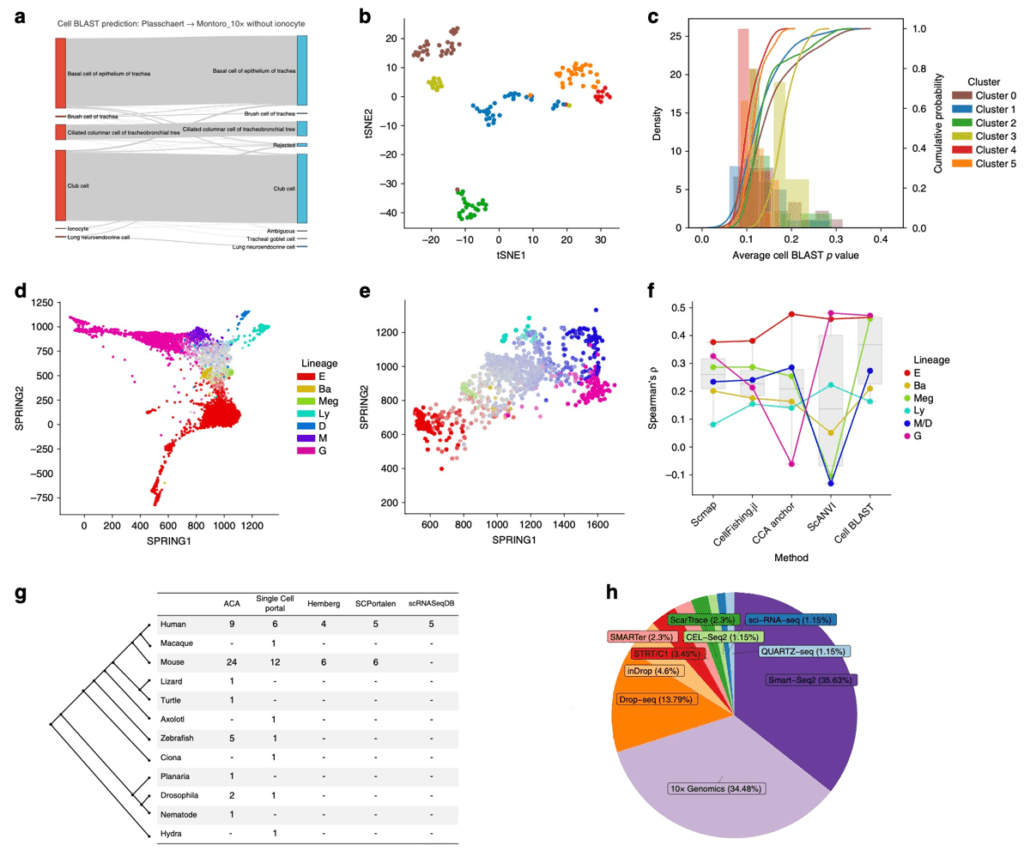
图3:Cell BLAST应用实例。(a) 用一个移除ionocyte的呼吸道数据集来注释另一个完整数据集的预测结果;(b) 检索过程中被reject细胞的聚类;(c) 各被reject细胞聚类的P-value分布,其中P-value最大(最不像reference)的一类即ionocyte;(d) 小鼠造血干细胞分化数据集的可视化(颜色代表分化命运,饱和度代表分化进程);(e) 利用小鼠造血干细胞分化数据来注释人造血干细胞分化数据;(f) 不同计算方法在跨物种分化命运预测效果上的比较;(g) ACA中不同物种数据集的数量与其他数据库的比较;(h) ACA中不同实验方法的数据量比较。
与序列BLAST一样,Cell BLAST要发挥实际作用,同样需要注释完备的大规模数据库作为支撑。通过收集大量已发表的单细胞转录组数据,我们建立了一个涵盖2,989,582个单细胞、8个物种、27个不同的组织器官的数据库,称为Animal Cell Atlas (ACA)(图3g, h)。我们对ACA中的细胞注释进行了详细的整理,并使用Cell Ontology构建了一套结构化的细胞类型标注,用于统一不同数据集中的标注以及支持细胞类型的推断。
Cell BLAST提供了在线检索平台(https://cblast.gao-lab.org),用户可以直接上传待注释的单细胞转录组数据,用ACA中的参考数据集进行细胞检索和自动注释;同时也提供了基于Python的开源软件包Cell BLAST(https://github.com/gao-lab/Cell_BLAST),用户可以使用软件包在自定义的参考数据集上进行模型训练、检索和定制化分析。
相关论文:
相关网站:
- Web Server: https://cblast.gao-lab.org
- Source Code: https://github.com/gao-lab/Cell_BLAST
参考文献:
- Tzeng E, Hoffman J, Saenko K, Darrell T. Adversarial discriminative domain adaptation. In IEEE Conference on Computer Vision and Pattern Recognition. 7167-7176 (2017).
- Montoro DT, et al. A revised airway epithelial hierarchy includes CFTR-expressing ionocytes. Nature 560, 319-324 (2018).
- Plasschaert LW, et al. A single-cell atlas of the airway epithelium reveals the CFTR-rich pulmonary ionocyte. Nature 560, 377-381 (2018).
- Tusi BK, et al. Population snapshots predict early haematopoietic and erythroid hierarchies. Nature 555, 54-60 (2018).
- Velten L, et al. Human haematopoietic stem cell lineage commitment is a continuous process. Nat Cell Biol 19, 271-281 (2017).