长非编码RNA(Long noncoding RNAs,lncRNA)是重要的调控分子,在多种生物学过程中发挥着调控作用(1)。多种生物学特征可用于研究长非编码RNA的功能及其潜在的机制。例如,研究长非编码RNA受到的自然选择可以揭示它们是否具有保守的功能(2);研究亚细胞定位可以揭示它们在何处发挥功能(3);通过对共表达的蛋白编码基因进行功能富集可以揭示它们发挥怎样的功能(4);研究相互作用的生物分子,如miRNA和RNA结合蛋白,可揭示潜在的功能机制(5, 6)。尽管已有部分数据库可为已知的长非编码RNA提供全面的注释(如LncBook (7),NONCODE (8)等),然而随着越来越多的新的长非编码RNA被鉴定出来,一个高效全面的注释工具仍然是重要的需求。
北京大学生物医学前沿创新中心 (BIOPIC)、生物信息中心(CBI)暨北京未来基因诊断高精尖创新中心(ICG)高歌课题组通过整合大量注释资源和分析工具,对课题组之前开发的人类长非编码RNA注释平台AnnoLnc (9)进行了更新。更新后的注释平台AnnoLnc2(http://annolnc.gao-lab.org/)目前可支持对人(参考基因组版本为hg38)和小鼠(参考基因组版本为mm10)的任意长非编码RNA序列进行在线注释。AnnoLnc2整合了十个注释模块(图1),涵盖序列和结构、表达和调控、功能和相互作用、以及演化和遗传关联,为研究长非编码RNA的功能及其作用机制提供了高效全面的分析平台。
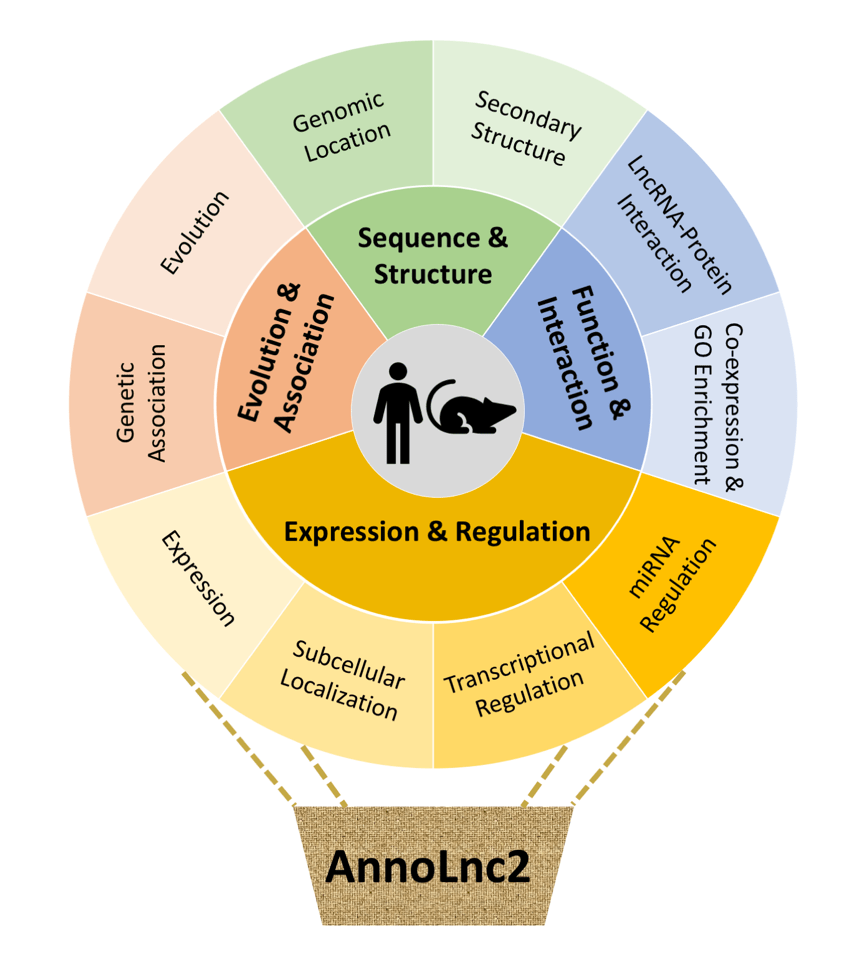
图1. AnnoLnc2框架
相比于AnnoLnc,AnnoLnc2整合了更多的注释资源。其中,表达模块可支持对目标序列在96个人类RNA-seq样本(涵盖26个组织,39个癌细胞系和2个正常细胞系)或69个小鼠RNA-seq样本(涵盖28个组织和7个细胞系)中的表达进行实时计算;转录调控模块整合了来自1339个人类转录因子和738个小鼠转录因子的结合位点;蛋白相互作用模块整合了来自188个人类RNA结合蛋白和62个小鼠RNA结合蛋白的结合位点。此外,AnnoLnc2还整合了一个新的注释模块-亚细胞定位,该模块可报告长非编码RNA的核质定位,以及已知的定位信号在目标序列中的分布。详细的数据介绍可参见http://annolnc.gao-lab.org/help.php#link-update。
AnnoLnc2网站操作便捷,用户仅需上传或者输入待分析的序列,选择相应物种,即可进行一键式分析(图2A)。每条序列的注释结果均有一个对应的网页界面,各模块的注释结果以交互式的图表的形式展示,以便用户检索感兴趣的条目。 用户可批量下载所有的注释结果(图2B)以用于后续研究。此外,AnnoLnc2还提供了单机版本(下载链接:http://annolnc.gao-lab.org/download.php),以便用户进行大规模的线下分析。
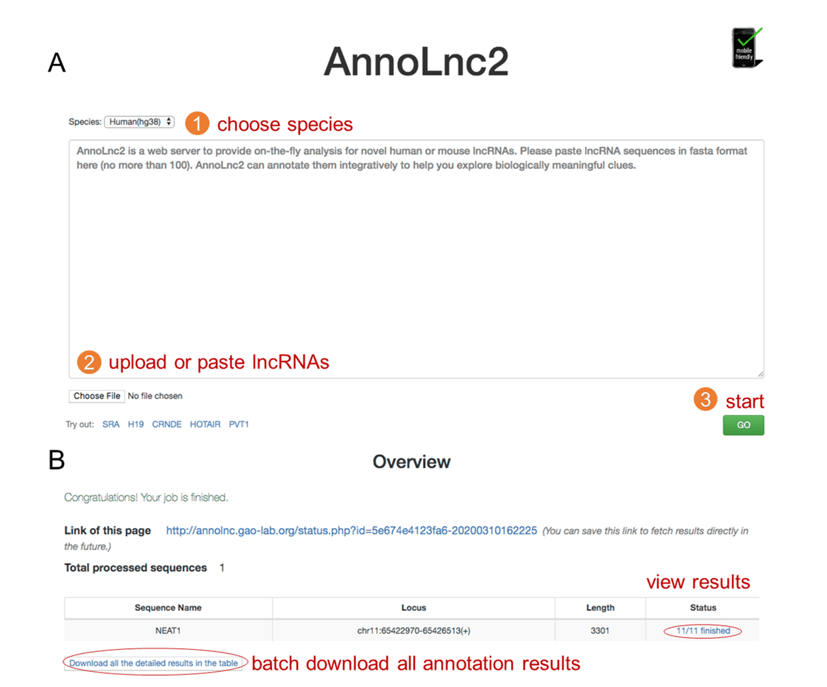
图2. AnnoLnc2 网页界面。用户可通过一个三步骤的操作运行AnnoLnc2(A),并查看详细的注释结果,以及批量下载所有的注释结果(B)。
该研究于2020年5月14日以AnnoLnc2: the one-stop portal to systematically annotate novel lncRNAs for human and mouse为题在线发表于Nucleic Acids Research。博士生柯岚、杨德昌为共同第一作者,高歌研究员为通讯作者。王宇、丁阳等合作者在平台构建和文章写作方面提供了大力支持。
AnnoLnc2网址:http://annolnc.gao-lab.org/
原文链接:https://doi.org/10.1093/nar/gkaa368
参考文献
- Marchese,F.P., Raimondi,I. and Huarte,M. (2017) The multidimensional mechanisms of long noncoding RNA function. Genome Biol., 18, 206.
- Ponjavic,J., Ponting,C.P. and Lunter,G. (2007) Functionality or transcriptional noise? Evidence for selection within long noncoding RNAs. Genome Res., 17, 556–65.
- Zhang,K., Shi,Z.M., Chang,Y.N., Hu,Z.M., Qi,H.X. and Hong,W. (2014) The ways of action of long non-coding RNAs in cytoplasm and nucleus. Gene, 547, 1–9.
- Guttman,M., Amit,I., Garber,M., French,C., Lin,M.F., Feldser,D., Huarte,M., Zuk,O., Carey,B.W., Cassady,J.P., et al. (2009) Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals. Nature, 458, 223–7.
- Yoon,J.-H., Abdelmohsen,K. and Gorospe,M. (2014) Functional interactions among microRNAs and long noncoding RNAs. Semin. Cell Dev. Biol., 34, 9–14.
- Castello,A., Fischer,B., Eichelbaum,K., Horos,R., Beckmann,B.M., Strein,C., Davey,N.E., Humphreys,D.T., Preiss,T., Steinmetz,L.M., et al. (2012) Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell, 149, 1393–406.
- Ma,L., Cao,J., Liu,L., Du,Q., Li,Z., Zou,D., Bajic,V.B. and Zhang,Z. (2019) LncBook: a curated knowledgebase of human long non-coding RNAs. Nucleic Acids Res., 47, 2699.
- Fang,S., Zhang,L., Guo,J., Niu,Y., Wu,Y., Li,H., Zhao,L., Li,X., Teng,X., Sun,X., et al. (2018) NONCODEV5: a comprehensive annotation database for long non-coding RNAs. Nucleic Acids Res., 46, D308–D314.
- Hou,M., Tang,X., Tian,F., Shi,F., Liu,F. and Gao,G. (2016) AnnoLnc: a web server for systematically annotating novel human lncRNAs. BMC Genomics, 17, 931.