长非编码RNA(Long noncoding RNAs,lncRNA)一般指长度大于200bp且不具有编码蛋白质功能的RNA(1,2)。长非编码RNA是重要的调控分子,在发育和疾病(3)等多种过程中发挥着重要的调控作用,有望成为药物靶标和疾病诊断的标志物(4)。尽管报道的人类长非编码RNA数量很多,但各个长非编码RNA参考注释集合之间存在很大差异,目前人类长非编码RNA注释还不完整。缺乏一个完整的、高质量的人类长非编码RNA参考注释集已经阻碍了对长非编码RNA转录调控系统结构与功能的深入理解。
北京大学生物医学前沿创新中心 (BIOPIC)、生物信息中心(CBI)暨北京未来基因诊断高精尖创新中心(ICG)高歌课题组通过整合大量样本资源,基于RNA-Seq数据构建了高质量的人类长非编码RNA参考注释集:RefLnc (Reference catalog of LncRNA, http://reflnc.gao-lab.org/)。RefLnc整合了来自30个人类正常组织、两个细胞系和18个肿瘤的14,166个poly-A + RNA-Seq样本信息,在正常组织中鉴定了27,520个新lncRNA(图1),显著扩展了目前人类lncRNA注释集。在此基础上,RefSeq系统注释了lncRNA在人类正常和癌症组织中的表达特征,并在全基因组范围探索了lncRNAs的生理功能和临床意义,为后续功能研究提供了重要的数据基础与线索。
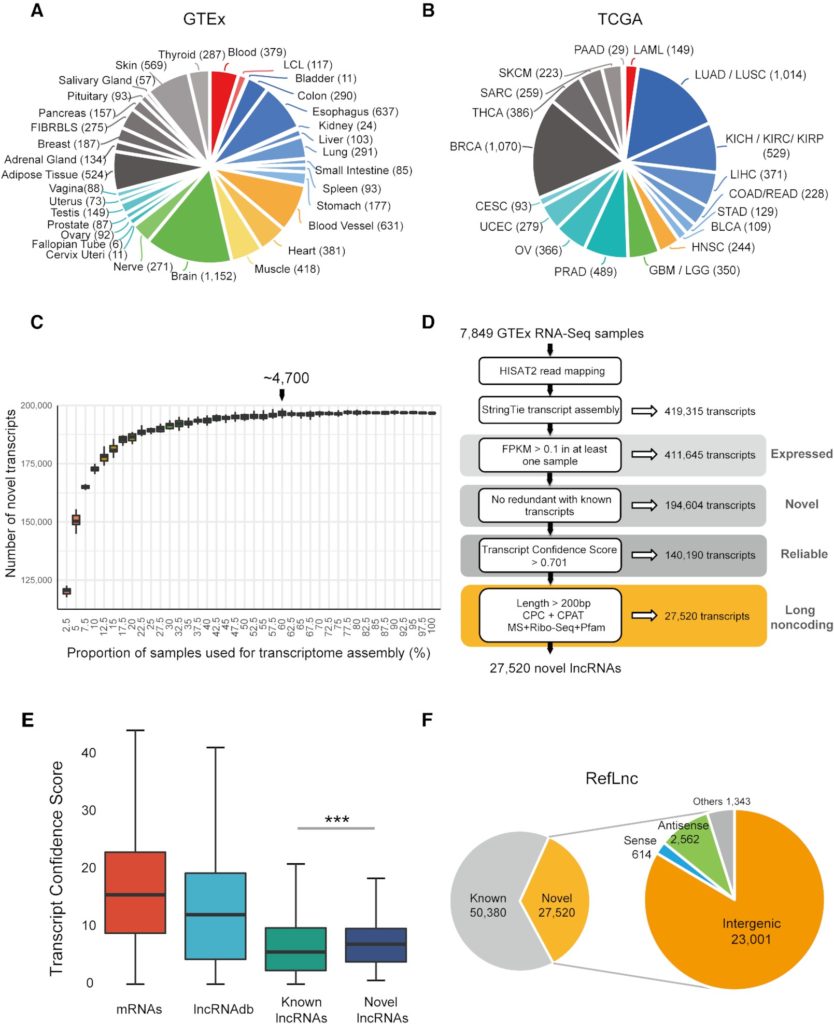
图1:RefLnc极大地扩展了人类lncRNA注释集。(A)来自GTEx的7,849个RNA-Seq样本被用于拼接转录本,其涵盖30个人类正常组织和两个细胞系。(B)来自TCGA中18个肿瘤的6,317个样本被用于分析转录本在肿瘤中的表达特性。(C)当RNA-Seq样本量接近4,700时,拼装出来的新转录本数量接近饱和。每个数据集包括所有类型的组织、性别和种族。(D)鉴定lncRNA的流程。(E)新lncRNA的转录本可靠分数(TCS)高于注释lncRNA。(F)RefLnc注释集含有77,900个lncRNA,包括经过验证的注释lncRNA和新lncRNA,并且83.6%的新lncRNA位于基因间区。
与先前的报道一致,与mRNA 相比,lncRNA转录本更短、外显子更少、GC含量更低、表达水平更低、剪切效率更低、保守性更低,且组织表达特异性更高(图2)。另外,此工作鉴定了75个新lincRNA在性别间差异表达(FDR<0.05)、132个表达与年龄相关(FDR <0.001),及70个在种族间差异表达(FDR <0.05)(图2)。种族差异的lincRNA更倾向于在脑(OR = 8.84, P = 3.05e–6)和睾丸(OR = 2.70, P = 1.21e–3)中表达。此外,160个新lincRNA与基因间区SNP位置重叠。例如,新lincRNA与甲状腺癌风险相关的SNP重叠(rs116909374)且这个lincRNA在甲状腺中特异性表达并在甲状腺肿瘤中显著上调(图2I)。
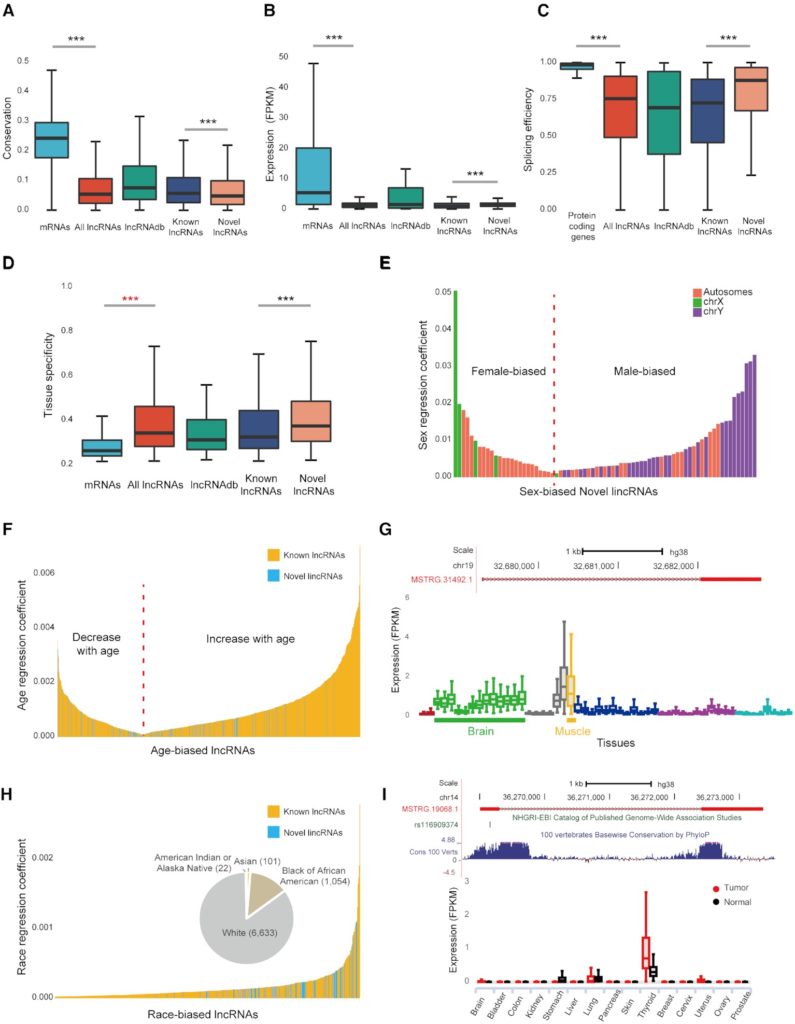
图2:RefLnc中lncRNA的特征。(A)lncRNAs的保守性低于mRNA。(B)lncRNA的表达水平低于mRNA的表达水平。(C)lncRNA可变剪接效率比蛋白质编码基因更低。(D)lncRNA的表达组织特异性比mRNA更高。(E)性别差异表达的新lincRNA(G)与年龄相关的新lincRNA MSTRG.31492.1在正常样本中的表达模式。(H)种族差异表达的新lincRNA(I)新lincRNA MSTRG.19068.1在肿瘤和正常组织间的差异表达模式,其与甲状腺癌风险相关的SNP位置重叠。
通过分析TCGA中18个肿瘤组织的6,317个样本,该工作鉴定了2,163个在肿瘤和正常组织之间差异表达的新lincRNA,且肿瘤特异性表达的新lincRNAs比例远高于注释lncRNAs和mRNA(图3)。此外,该工作鉴定了与临床结果(例如肿瘤转移、复发、临床分期和生存率等)相关的新lincRNA(图3C)。例如,180个新lincRNA与脑肿瘤的总体存活时间相关,其中约一半(47.2%,76/161)在独立胶质瘤样本中成功验证。
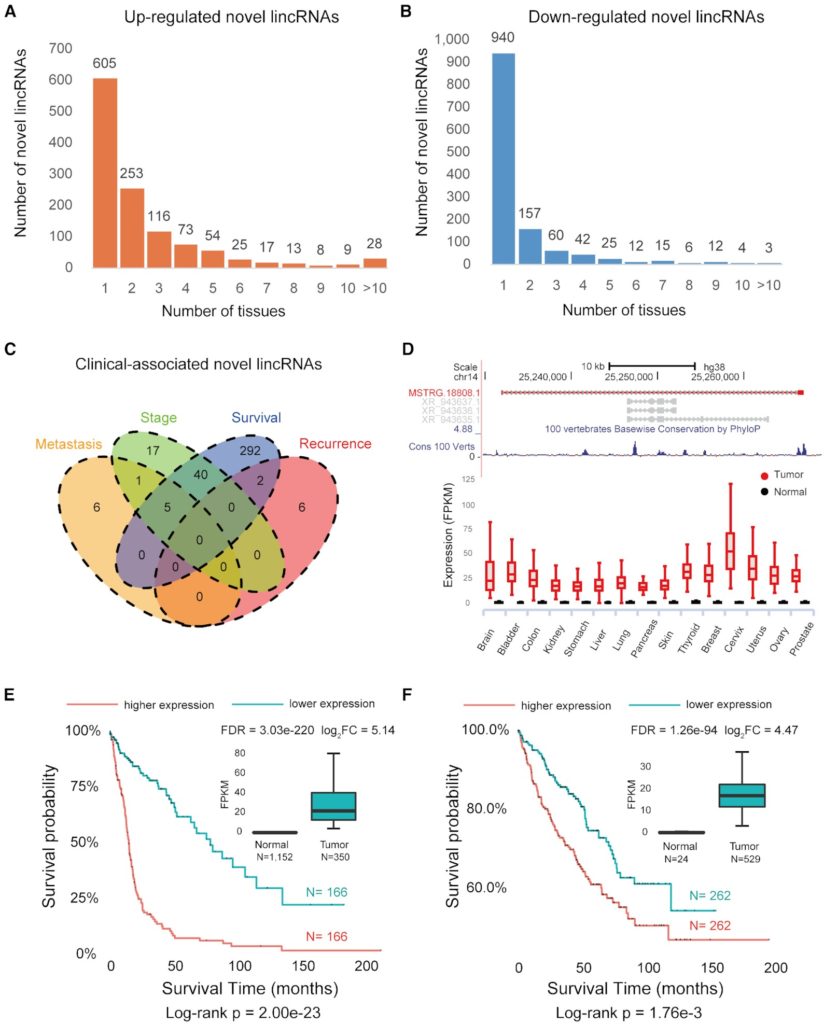
图3:鉴定与肿瘤相关的新lincRNA。(A)肿瘤中上调的新lincRNA。(B)肿瘤中下调的新lincRNA。(C)临床相关的新lincRNA。(D)MSTRG.18808.1在肿瘤和正常组织间差异表达模式。(E)MSTRG.18808.1的表达与脑肿瘤患者存活率相关。(F)MSTRG.18808.1的表达与肾肿瘤患者存活率相关。
该工作开发了RefLnc在线网站(http://reflnc.gao-lab.org/),对lncRNA在7,849个正常样本和6,317个肿瘤样本的详细表达特征进行系统注释(图4)。
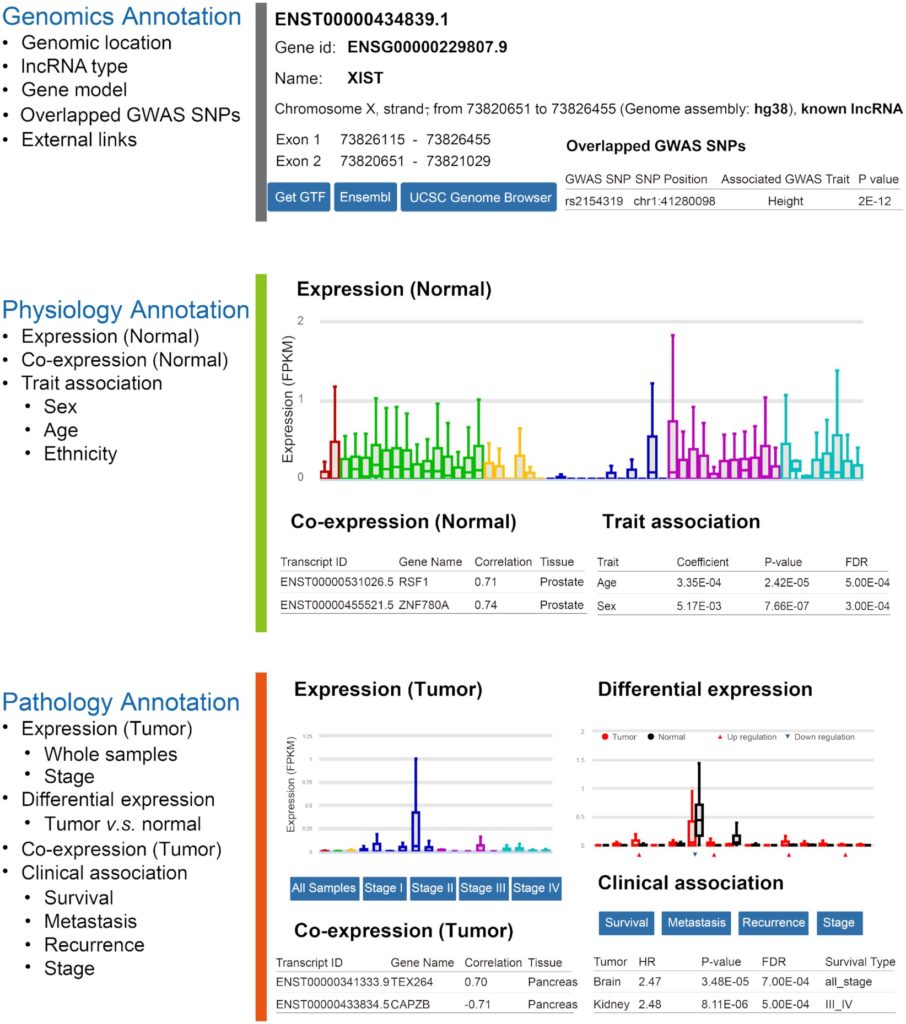
图4:RefLnc在线服务器。它提供了RefLnc中每个lncRNA的详细注释,包括基因组学注释、生理学注释和病理学注释。
该研究于2019年8月6日以An expanded landscape of human long noncoding RNA为题在线发表于Nucleic Acids Research。博士生降帅、程斯进为共同第一作者,高歌研究员为通讯作者。任立晨、王倩、亢雨笺、丁阳、侯玫、杨晓旭、林媛和梁楠等合作者在文章写作、实验验证、数据分析方面提供了大力支持。
Website: http://reflnc.gao-lab.org/
原文链接: https://academic.oup.com/nar/advance-article-abstract/doi/10.1093/nar/gkz621/5539882
参考文献:
- Kapranov, P., Cheng, J., Dike, S., Nix, D.A., Duttagupta, R., Willingham, A.T., Stadler, P.F., Hertel, J., Hackermuller, J., Hofacker, I.L. et al. (2007) RNA maps reveal new RNA classes and a possible function for pervasive transcription. Science, 316, 1484-1488.
- Mattick, J.S. and Rinn, J.L. (2015) Discovery and annotation of long noncoding RNAs. Nature structural & molecular biology, 22, 5-7.
- Batista, P.J. and Chang, H.Y. (2013) Long noncoding RNAs: cellular address codes in development and disease. Cell, 152, 1298-1307.
- Wahlestedt, C. (2013) Targeting long non-coding RNA to therapeutically upregulate gene expression. Nature Reviews. Drug discovery, 12, 433-446.